Gromacsを使ったペプチド鎖の分子動力学計算
Last Update:2021/12/09
Introduction
Gromacsは分子動力学シミュレーションを行うオープンソースソフトウェアである. タンパク質や核酸などの生体分子の動力学計算に便利な機能を多数有しており, 生物や製薬の分野でよく用いられている. タンパク質や核酸の構造データはデータベース(PDBなど)からダウンロードすることができる. これらの生体分子の構造データをもとに, 水などの溶媒分子を配置して, 分子動力学計算を実行する. このような分子動力学シミュレーションはタンパク質とリガンドの相互作用解析などに用いられ, さまざまな形で役立てられている. ここでは水中におけるペプチド鎖の分子動力学計算をMateriApps LIVE!上で行ってみよう.
本チュートリアルで使用したMateriApps LIVE!のバージョンは2.4である.
実行方法
ここではあらかじめ用意されたスクリプトファイルによって, [KIGAKI]3と呼ばれる小さなペプチド鎖の水中でのダイナミクスをシミュレーションする(詳細はGromacsTutorialを参照). 計算時間が長いため, あらかじめVirtualBoxの設定でCPU数をなるべく多くしておくとよい. MateriApps LIVE!を起動するまえに「設定>システム>プロセッサー」とすることでVirtualBox内で使用するCPUプロセッサの数を変更できる(PCのCPUコア数を調べ、それと同じにしておくとよい. 通常はCPU数選択画面で緑のバーの範囲内で最大に設定すればよいはずである.). 次にMateriApps LIVE!を起動・ログインし, 画面左下のスタートメニューから「System Tools>LXTerminal」を選択して端末を立ち上げる.端末で以下のコマンドをタイプし, 1行ごとに改行キーを押す:
$ wget https://github.com/cmsi/malive-tutorial/releases/download/em2020/gromacs.tgz $ tar zxvf gromacs.tgz $ cd gromacs $ sh run.sh
並列度はソフトウェア側で自動判定してくれる. この分子動力学計算はノートPCでは3〜5時間程度, 性能のよいデスクトップマシンなら1時間ほどで終了するはずである. 次に可視化のためにVMDというソフトをインストールしよう. MateriApps LIVE!では, VMDはライセンスの関係上, GUIによるセットアップツール(VMD Setup)のみが提供されている. まずVMDのWebページから「Download VMD」をクリックし, 適当なバージョン・OSを選択する. ユーザー名とパスワードの登録があるので, 登録のみを行う(ダウンロードが始まるかもしれないがこのファイルは不要). 次にMateirApps LIVE!上で「スタートメニュー>MateriApps>VMD Setup」を選択し, 現れたウィンドウで登録したユーザー名・パスワードを入力して「Install」ボタンを押す. これにより可視化ソフトVMDがインストールされるはずである. その後, 端末から下記のコマンドを実行する:
$ sh animation.sh
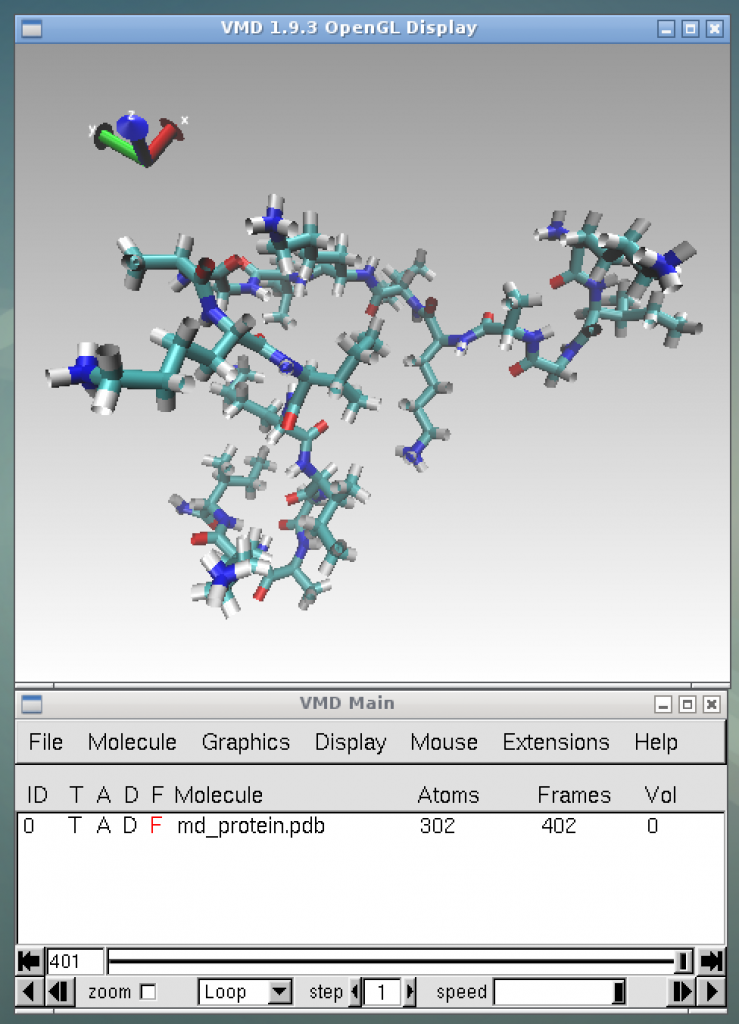
これにより, [KIGAKI]3の分子構造が出力されるはずである(下図). 2つのウィンドウのうち, 分子が表示されていないウィンドウの右下にある再生ボタンを押すと, 分子構造の時間発展がアニメーション表示される.
Gromacsの設定方法はやや複雑である. スクリプトファイルのrun.shの中身をみる(端末でmore run.shと入力して改行キーを押すと見ることができる)とわかるように様々なコマンドを打ち込むことで, 箱の作成や水の配置などを行っている. 元となる分子の構造ファイルはPDBファイル(拡張子は.pdb)によって与えられる. また加工した結果は様々なファイル(.gro, .topなどの拡張子をもつ)に書き込まれる(詳細はGromacsTutorialを参照). 分子動力学の計算条件はmdpファイルに入っている. ここでは詳細は書かないが, これらのコマンドや入力ファイルを準備することで細やかな設定が可能である.
おわりに
Gromacsにおいても力場の選択(スクリプトrun.sh中の一行目のコマンドで選択している)は重要である. さらにタンパク質の構造ファイル(PDBファイル)はPDBからダウンロードできるが, 一部の構造や水素原子が省略されている場合があり, しばしばそれらを補完する必要がある. 当然ながらタンパク質のような大きな分子の計算にはそれなりの計算リソースが必要であり, 仮想環境を利用しているMateriApps LIVE!では力不足である. とはいうものの, ある程度のスペックを持つPCもしくは計算機サーバに直接Gromacsをインストールすれば, 数時間から1日程度で分子動力学計算を終えることができる. かつてはスーパーコンピュータでしか取り扱えなかったタンパク質の動力学計算が, あらゆる研究者の手に届くところまできているのである!